Czy można ujarzmić ewolucję czyli słów kilka o ukierunkowanej ewolucji enzymów
Ewa Kot 1, Katarzyna Kurpiewska 2, Maciej Szaleniec 1
1 Instytut Katalizy i Fizykochemii Powierzchni im. Jerzego Habera Polskiej Akademii Nauk, Kraków
2 Wydział Chemii, Uniwersytet Jagielloński, Kraków
Streszczenie
Ewolucja jest niesamowitym procesem, który przez miliardy lat doprowadził do przeobrażenia prymitywnych komórek pierwotnego życia w cały niesamowicie skomplikowany i bioróżnorodny ekosystem naszej planety. W procesie ewolucji przeobrażeniom uległy wszystkie komponenty budujące komórki żywe, w tym również białkowe enzymy przyspieszające reakcje chemiczne. Obecnie enzymy coraz chętniej wykorzystujemy w przemyśle. Dzięki technikom biologii molekularnej i inżynierii genetycznej możliwe stało się ich modyfikowanie dzięki czemu uzyskują one zdolność do katalizowania przemian chemicznych, które nie mają swojego odpowiednika w organizmach żywych. W chwili obecnej jedną z bardziej obiecujących metod takich modyfikacji jest ukierunkowana ewolucja enzymów. W uznaniu przełomowego znaczenia molekularnych technik ewolucyjnych dla Nauki 3 października 2018 roku Królewska Szwedzka Akademia Nauk ogłosiła zwycięzców tegorocznej Nagrody Nobla z Chemii. Nagrodę, przyznaną za “wykorzystanie mechanizmów ewolucji”, podzielono na dwie części – pierwszą część przyznając pani Frances H. Arnold z Caltech w Kalifornii, drugą zaś Georgowi Smithowi z University of Missouri w stanie Kolumbia oraz Sir Gregorowi P. Winterowi z MRC Laboratory of Molecular Biology w Cambridge w Wielkiej Brytanii. Metoda ukierunkowanej ewolucji enzymów rozwinięta przez profesor Arnold, piątą kobietę w historii uhonorowaną Nagrodą Nobla, została przybliżona w niniejszym artykule.
3 października 2018 roku Królewska Szwedzka Akademia Nauk ogłosiła zwycięzców tegorocznej Nagrody Nobla z Chemii. Nagrodę, przyznaną za “wykorzystanie mechanizmów ewolucji”, podzielono na dwie części – pierwszą część przyznając pani Frances H. Arnold z Caltech w Kalifornii, drugą zaś Georgowi Smithowi z University of Missouri w stanie Kolumbia oraz Sir Gregorgowi P. Winterowi z MRC Laboratory of Molecular Biology w Cambridge w Wielkiej Brytanii. Pierwsza część Nagrody dotyczyła badań nad kierowaną ewolucją enzymów. Metoda rozwinięta przez profesor Arnold, piątą kobietę w historii uhonorowaną Nagrodą Nobla, w ciągu ostatnich dwóch dekad dokonała przełomu w dziedzinie przemysłowego zastosowania enzymów. I właśnie tę rewolucyjną metodę przybliża niniejszy artykuł.
Enzymy są białkami, które w organizmach żywych odpowiedzialne są za przyspieszanie i kontrolowanie większości procesów chemicznych. Szersze omówienie zagadnienia jakim są enzymy można znaleźć w jednym z poprzednich numerów Wszechświata (Wszechświat, 2017, t. 118, strony 108 – 117). Obecnie są chętnie wykorzystywane w przemyśle jako biokatalizatory reakcji chemicznych ze względu na znacznie większą wydajność i selektywność katalizowanych przez nie przemian w stosunku do konwencjonalnie wykorzystywanych katalizatorów. Problem jednak stanowi fakt, że tylko niewielka część enzymów występujących w naturze posiada cechy, które umożliwiają ich przemysłowe wykorzystanie np. odporność na wysoką temperaturę, obecność rozpuszczalników organicznych, czy też możliwość wykorzystania tej samej porcji enzymu w kilku cyklach reakcji [9]. Bardzo często chcemy również wykorzystać enzymy do katalizowania przemiany nietypowych cząsteczek chemicznych, które nie mają swojego odpowiednika w organizmach żywych – niestety naturalne enzymy takich cząsteczek nie rozpoznają! Najbardziej bezpośrednim sposobem uzyskania enzymów o pożądanych właściwościach byłoby tzw. racjonalne przeprojektowanie białek, czyli taka modyfikacja ich budowy na poziomie ich sekwencji aminokwasowej, która pozwoli na uzyskanie pożądanych cech. Do racjonalnego przeprojektowania niezbędna jest jednak wiedza o zależnościach pomiędzy sekwencją aminokwasową zapisaną w genach niosących informację o enzymie, a jego strukturą i właściwościami katalitycznymi. Znajomość tych zależności jest dostępna jedynie dla niewielkiego procenta enzymów, które mogą być użyteczne w przemyśle [7]. Rozwiązaniem jest zatem technika, która pozwala na modyfikacje białek w pożądanym kierunku bez znajomości wspomnianych zależności. Jedną z najbardziej skutecznych technik takich zmian jest właśnie tytułowa „ukierunkowana ewolucja enzymów”.
Od DNA do enzymu
Zanim przejdziemy do wyjaśniania tajników kierowanej ewolucji enzymów, trzeba wyjaśnić kilka podstawowych pojęć biologii molekularnej. Enzymy są białkami, które w organizmach żywych odpowiedzialne są za przyspieszanie i kontrolowanie większości procesów chemicznych. Centralny dogmat biologii molekularnej, czyli teoria, która została przyjęta bezwarunkowo i nie podlega podważeniu, zakłada, że białka są ostateczną fizyczną postacią informacji zawartej w DNA (kwasie deoksyrybonukleinowym) (Ryc. 1). Z grubsza można by to porównać do wypieku ciasta. Ciastko jest fizycznym odzwierciedleniem informacji podanych w przepisie na jego wykonanie. Genetyczny przepis na życie zapisany w postaci DNA jest zawarty we wszystkich organizmach na Ziemi i jest on przekazywany pokoleniowo, czyli np. rodzice przekazują materiał swoim dzieciom, dzieci wnukom itd. W materiale są zapisane między innymi takie informacje jak np. kolor oczu czy włosów. DNA ma formę dwóch nici, na której, jak na taśmie magnetycznej, zapisany jest przepis jak wytworzyć wszystkie białka potrzebne do budowy danej komórki. Na każdej z nici ,,zawieszone” są 4 rodzaje cegiełek/liter kodu genetycznego zwanych nukleotydami: adenozyna, cytozyna, guanina i tymina. Nici DNA splecione są ze sobą tworząc specyficzną spiralę określaną mianem helisy. Cegiełki zawieszone na obu niciach muszą być do siebie ściśle dopasowane tworząc pary adenozyna – tymina, guanina – cytozyna. Kolejność ułożenia tych par nukleotydów nazywana jest sekwencją DNA. Powstanie białka z informacji zapisanych w DNA określa się mianem jego ekspresji, a od DNA do białka prowadzą 2 podstawowe procesy: transkrypcja i translacja.

Ryc. 1 Struktura DNA i schemat transkrypcji DNA na mRNA a następnie translacji mRNA na polipeptyd.
W trakcie transkrypcji informacja zapisana na DNA jest przepisywana na RNA (tzw. mRNA). RNA (kwas rybonukleinowy) jest w przeciwieństwie do DNA jednoniciowy i zamiast tyminy w swoim zestawie genetycznych liter wykorzystuje uracyl. W trakcie transkrypcji helisa DNA ulega chwilowemu rozpleceniu. Jedna z nici stanowi matrycę, czyli podstawę, na bazie której dobudowywana jest nić RNA, której nukleotydy tworzą komplementarne połączenia z nukleotydami na nici DNA. I tak jeśli na nici DNA mamy ciąg adenina-guanina-cytozyna-tymina-guanina to komplementarna do tego nić RNA będzie posiadała ciąg uracyl-cytozyna-guanina-adenina-cytozyna. Po przepisaniu całej sekwencji z DNA na RNA, helisa ponownie ulega spleceniu do swojej pierwotnej formy, a RNA jest zaangażowane w dalsze etapy produkcji białka. Sekwencja cegiełek/liter w RNA jest przepisem na sekwencję aminokwasów w białku w procesie określonym mianem translacji.
W trakcie syntezy białka nukleotydy RNA są czytane trójkami, trzy zasady RNA kodują jeden aminokwas w białku. Taką trójkę zasad RNA określa się mianem kodonu i możemy ją sobie wyobrazić jako pojedyncze słowo w przepisie. Kodem genetycznym zaś nazywamy reguły tłumaczenia sekwencji nukleotydów (i zbudowanych z nich słów-kodonów) w kwasie nukleinowym (DNA czy też RNA) na sekwencję aminokwasów w białku określana. Mamy 61 możliwych kodonów, przy czym każda trójka nukleotydów może kodować tylko jeden aminokwas. Po zakończeniu translacji powstały peptyd (czyli łańcuch aminokwasów) może być poddany modyfikacjom, może być do niego dobudowywany inny peptyd, może też zostać przetransportowany do docelowego miejsca w komórce, w którym to ma pełnić nadane mu funkcje. Słowem może stanowić element budulcowy komórki, stać się cząsteczką sygnałową bądź też przyjąć rolę katalizatora konkretnego procesu, czyli przyjąć formę enzymu [2].
Ukierunkowana ewolucja enzymów
Ewoluować od łacińskiego evolvere, oznacza dosłownie „rozwijać się”, „ujawniać nieodkryte dotąd możliwości” [8]. Od samego początku istnienia życia na Ziemi enzymy przechodzą proces ciągłych zmian pozwalający organizmom na adaptację do stale zmieniającego się środowiska. Źródłem tych zmian są mutacje, które są spowodowane przypadkowymi błędami, powstającymi w czasie kopiowania DNA podczas podziału komórek. Czynnikiem selekcyjnym jest zdolność potomnego organizmu do przeżycia – jeżeli wzrasta, dana mutacja ma szansę ulec propagacji (rozprzestrzenieniu się) w populacji. Ukierunkowana ewolucja enzymów nie jest niczym innym jak przeniesieniem procesów zachodzących w naturze do laboratorium i przyspieszeniem mających nastąpić zmian [11]. Pierwsze doniesienia użyteczności tej metody pojawiły się w ostatnich dekadach XX wieku. W 1984 roku Manfred Eigen opublikował pracę teoretyczną, w której opisał możliwy schemat pracy mający na celu skonstruowanie ,,maszyny ewolucyjnej” służącej do optymalizacji już istniejących enzymów [6].
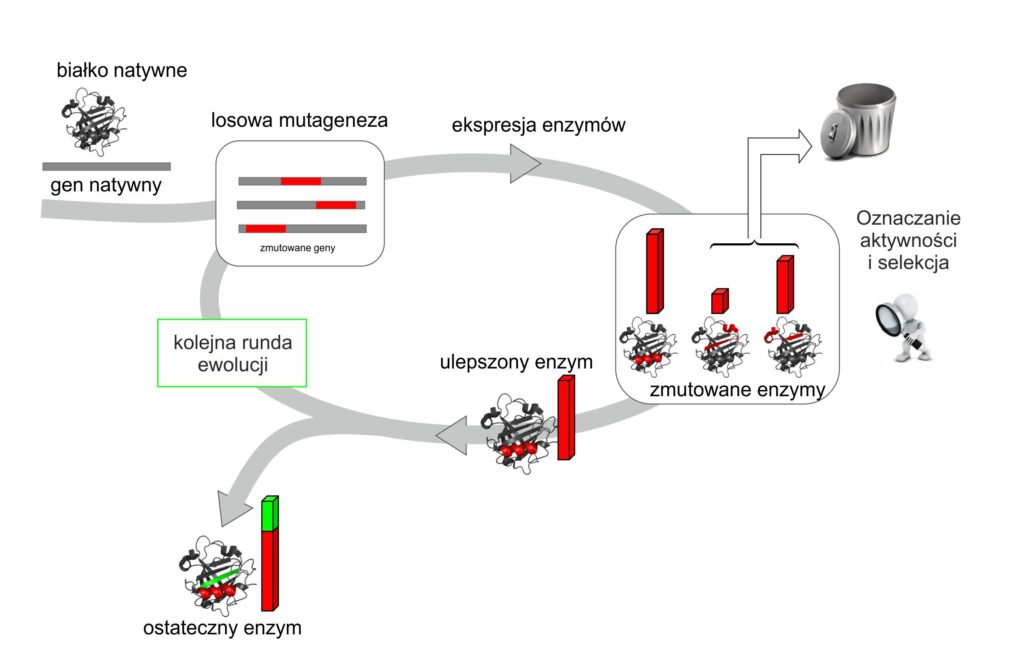
Ryc. 2. Schemat przedstawiający zasadę kierowanej ewolucji enzymów.
Strategia ukierunkowanej ewolucji opiera się na izolacji genu kodującego dany enzym, a następnie jego kopiowaniu z wprowadzeniem pewnej zmienności na poziomie kodu genetycznego (czyli sprowokowania mutacji) (Ryc. 2). W wyniku takiej podatnej na mutacje metody powstaje bardzo wiele zmienionych kopii oryginalnego genu. Wprowadzone zmiany w genie skutkują modyfikacją sekwencji aminokwasowej białka enzymatycznego stanowiącego przedmiot zainteresowania, co z kolei prowadzi do zmian na poziomie jego struktury i funkcji. Pozwala to na stworzenie pewnego rodzaju biblioteki sekwencji kodujących różne warianty tego samego enzymu. Odnosząc się do porównania z początku artykułu, do przepisu na ciasto zostają wprowadzone pewne modyfikacje, np. zamiast 100 gramów mąki pszennej mamy 130 gramów, zamiast 50 ml mleka dodamy 70 ml, albo odrobinę śmietany itp. Modyfikacja nie pozostanie bez wpływu na strukturę ciastka. Będzie ono bardziej zbite poprzez zwiększoną ilość mąki, bardziej wilgotne poprzez dodanie większej ilości mleka czy bardziej aromatyczne od śmietany, wszak tłuszcz zawarty w śmietanie jest nośnikiem smaku. Należy jednak podkreślić, że zmienność sekwencji białek w przypadku ukierunkowanej ewolucji jest losowa, w przeciwieństwie do racjonalnego i ukierunkowanego przeprojektowania, gdzie zmianie podlegają racjonalnie wybrane aminokwasy budujące białko. Oznacza to, że w przypadku racjonalnego przeprojektowywania wiemy, że chcemy dostać ciastko bardziej wilgotne więc zwiększamy ilość mleka, natomiast w przypadku ukierunkowanej ewolucji tworzymy zbiór przepisów, w których losowo zmieniamy ilość składników tworzących przepis i sprawdzamy jak nowy skład wpływa na wygląd, strukturę, smak ciastka. W dalszych etapach losowo zmienione sekwencje DNA są wprowadzane do komórek, bakteryjnych bądź eukariotycznych. Komórki są następnie hodowane, celem sprawdzenia, czy np. nowy wariant enzymu nie spowodował destrukcyjnych zmian prowadzących np. do braku wzrostu takiej komórki. Komórki, które przeżyły są następnie testowane pod względem ekspresji zmienionych sekwencji DNA, tak by sprawdzić czy wprowadzone mutacje nie spowodowały np. zablokowania syntezy nowej formy enzymu. W etapie końcowym uzyskujemy bibliotekę komórek, które posiadają podlegające ekspresji różne warianty, tzw. mutanty, enzymu. Zbiór komórek nazywamy biblioteką, bo każda z nich zawiera trochę inny przepis na nasz enzym. Następnie wykonywany jest proces selekcji, prowadzący do wytypowania organizmów posiadających enzym o ulepszonych właściwościach (np. poprzez zbadanie odporności enzymu na wysoką temperaturę albo zdolności do katalizowania reakcji z nowym substratem). Zidentyfikowanie tych wariantów genów, które kodują “ulepszone” enzymy pozwala na ich dalsze wykorzystanie. Zwykle przeprowadza się kolejne rundy losowej mutagenezy i selekcji, które skutkują nagromadzeniem pożądanych cech w jednym organizmie [1].
Po około 10 latach od ukazania się teoretycznego artykułu Eigena w 1993 roku pojawiła się pierwsza praca eksperymentalna autorstwa Frances H. Arnold, w której autorka opisała otrzymany przy pomocy ukierunkowanej ewolucji zmodyfikowany wariant enzymu subtylizyny E. Naturalna subtylizyna E jest enzymem, który katalizuje reakcje hydrolizy białek wyłącznie w wysoce zasadowym środowisku. Mówiąc prościej enzym ten katalizuje rozcięcia zbudowanego z aminokwasów łańcucha peptydowego w innych białkach obecnych w komórce. Należy też dodać, że naturalna subtylizyna E nie wykazuje aktywności katalitycznej w obecności rozpuszczalników organicznym, co znacznie utrudnia jej zastosowanie w syntezie organicznej. Po przeprowadzeniu kilku cykli ukierunkowanej ewolucji Arnold otrzymała nowy typ enzymu, który w obecności 60% dimetyloformamidu wykazuje 256-krotnie wyższą aktywność w porównaniu do jego formy naturalnie występującej w środowisku [5, 9]. Arnold opracowała cały schemat ukierunkowanej ewolucji obejmujący etapy identyfikacji enzymu mającego podlegać temu procesowi, techniki wprowadzania losowych mutacji do sekwencji enzymu, otrzymywanie bibliotek różnych jego wariantów oraz dobór warunków selekcji i wybór najbardziej korzystnych form badanego enzymu.
Laboratoryjna ewolucja w służbie przemysłu
Od tamtej pory naukowcy coraz chętniej korzystają z metod ukierunkowanej ewolucji co zdecydowanie poszerza wachlarz możliwości wykorzystania znanych enzymów jako skutecznych biokatalizatorów. Na całym świecie z zastosowaniem tej metody od lat opracowywane są enzymy stanowiące wydajną i przyjazną środowisku alternatywę dla używanych do tej pory katalizatorów. Warianty enzymów powstałe w wyniku ukierunkowanej ewolucji znalazły swoje zastosowanie nie tylko w wielu dziedzinach przemysłu (np. przemyśle spożywczym, tekstylnym, chemicznym czy biomedycznym), ale także w gospodarstwie domowym (np. enzymatyczne proszki do prania).
Dobrym przykładem takich enzymów są celulazy, które znalazły zastosowanie zarówno w przemyśle tekstylnym jak i gospodarstwie domowym. Wykorzystywane są m.in. w końcowych etapach powstawania dżinsu, do zmiękczania bawełny, czy też w detergentach jako składniki chroniące kolor. Celulazy wykorzystywane jako składnik detergentów powinny charakteryzować się kompatybilnością z innymi komponentami płynu czy proszku do prania, stabilnością termiczną i aktywnością w warunkach zasadowych. Sangren ze współpracownikami poprawili stabilność temperaturową jednej z celulaz o 7.7°C, czyli używając detergentu zawierającego ten enzym możliwe jest jego zastosowanie w wyższej temperaturze namaczania, czy też prania [10].
Jednym z bardziej współczesnych wyzwań nauki jest zrównoważona i przyjazna dla środowiska produkcja biopaliw mogących stanowić alternatywę dla paliw kopalnych. Dąży się do wytwarzania gotowych komponentów paliwa lub cząsteczek budulcowych z których przemysł petrochemiczny może paliwa zsyntetyzować z surowców odnawialnych. Jednym z potencjalnych kandydatów do takiej roli jest izobutanol, który może zarówno stanowić dodatek do benzyny jak i zostać przetworzony na krótkołańcuchowe alkany. Izobutanol może być produkowany na drodze biosyntezy z cukrów w rekombinowanych (tzn. zmodyfikowanych genetycznie) komórkach E. coli. Jednakże dwa enzymy szlaku zaangażowanego w jego syntezę wymagają obecności specyficznego kofaktora tzw. NADPH (fosforan dinukleotydu nikotynamidoadeninowego). Niestety glikoliza, która jest naturalnym procesem towarzyszącym wzrostowi bakterii, powoduje powstawanie dużych ilości innego kofaktora, NADH (dinukleotyd nikotynoamidoadeninowy). Arnold i współpracownicy zastosowali ukierunkowaną ewolucję by zmienić zależność obu enzymów szlaku biosyntezy od NADPH na zależność od NADH, co umożliwiło produkcję izobutanolu w komórkach bakteryjnych [3].
Innym przykładem zastosowania ukierunkowanej ewolucji na pograniczu biotechnologii i biomedycyny jest opracowanie strategii znakowania zbliżeniowego białek (ang. proximity labeling). Znakowanie zbliżeniowa to jedna z technik analizy proteomicznej białek. Proteomika to gałąź nauki, która zajmuje się mówiąc najogólniej wszystkim co jest związane z białkami, a co za tym idzie analizą struktury białek, pełnionych przez nie funkcji, odkrywaniem zależności jakie zachodzą między białkami czy też między białkami a innymi elementami w komórce i organizmie żywym. Technika znakowania zbliżeniowego umożliwia wgląd w skomplikowane procesy zachodzące w komórce. Jak jednak właściwie ona działa? Pierwszym krokiem jest genetyczne zmodyfikowanie genów badanego białka (białko A) w organizmie żywym. Modyfikacja wytwarza tzw. białko fuzyjne, czyli „dokleja” do badanego białka A specjalny enzym (E), który katalizuje reakcję znakowania innych białek. W wyniku ekspresji powstałego białka fuzyjnego (AE) w miejscu, gdzie normalnie w komórce przebywa badane przez nas białko A, rozpoczyna się proces znakowania wszystkich innych białek, które zbliżą się na niewielką (nanometryczną) odległość do białka fuzyjnego AE. W kolejnym kroku zaznaczone przez enzym E białka można łatwo wyizolować z komórki i zidentyfikować. Dzięki tej technice możemy dowiedzieć się jakie białka są produkowane w organellach komórkowych lub określić z jakimi białkami oddziałuje śledzone przez badacza białko A. Te informacje można wykorzystać do lepszego projektowania leków lub testów wykrywających choroby metaboliczne, w których duże znaczenie mają enzymy występujące w organizmie człowieka.
W technice tej stosuje się dwa enzymy zdolne do znakowania białek za pomocą biotyny, peroksydazę askorbinianu wyizolowaną z soi (zwana dalej APEX2) oraz ligazę biotyny wyizolowaną z E. coli (zwana dalej BiolD). APEX2 jest enzymem, który pozwala na bardzo szybkie znakowanie znajdujących się w pobliżu białek, a co za tym idzie na identyfikację dynamicznych interakcji zachodzących w żywych komórkach. Niestety jego wykorzystanie wiąże się z koniecznością użycia nadtlenku wodoru (wody utlenionej). Odczynnik ten po pierwsze jest toksyczny, a po drugie trudny do dostarczenia do wnętrza komórek bez ich uszkodzenia.
Z kolei BiolD jest atrakcyjnym enzymem ze względu na prostotę znakowania oraz nietoksyczne warunki tego procesu. Niestety, w przeciwieństwie do APEX2, znakowanie wykorzystujące BiolD wymaga znacznie więcej czasu, bo od 18 do 24 godzin, co kompletnie uniemożliwia obserwację dynamicznych procesów zachodzących w komórkach. Brannon i współpracownicy opracowali dwa nowe warianty ligazy biotyny tj. TurbolD oraz miniTurbo, dzięki którym udało się skrócić proces znakowania z wielu godzin do około 10 minut. Możliwe stało się również wykorzystanie tego typu znakowania w komórkach drożdży (a więc w komórkach eukariotycznych, podobnych do naszych), co wcześniej było niemożliwe [4].
Ukierunkowana ewolucja stała się także pomysłem na biznes. Na światowych rynkach istnieje kilka firm, które specjalizują się w poprawianiu enzymów już wykorzystywanych w przemyśle oraz tworzenia nowych białek mogących mieć w komercyjne zastosowanie. Do takich firm zaliczyć można np. ATUM Company, serwis GeneArt Directed Evolution działający pod firmą ThermoFischer Scientific, czy też Creative Enzymes [12 – 14].
Podsumowanie
Minęło prawie 35 lat od okrycia metody ukierunkowanej ewolucji enzymów. Chociaż metoda ta pozwala dziś na tworzenie enzymów zdolnych do jeszcze szybszego katalizowania reakcji czy też zmiany możliwych warunków, w których dana reakcja może zachodzić, bez konieczności pełnego zrozumienia zależności między strukturą a funkcją enzymu wciąż jednak jest mnóstwo miejsca na kolejne odkrycia. Gdy dziennikarze zapytali profesor Arnold jakie ma rady dla osób, które dążą do osiągnięcia wielkich rzeczy odpowiedziała, że najważniejsze by lubić to nad czym się pracuje, dzień za dniem i by ryzykować, bo jeszcze nikt, kto osiągnął sukces nie postępował zgodnie z ,,przepisem”.
Bibliografia:
- Arnold FH. (2018). Enzymes by Evolution: Bringing New Chemistry to Life. Molecular Frontiers Journal 2: 9 – 18.
- Barciszewski, J; Markiewicz, WT. (2002). “Kwasy nukleinowe. Kod genetyczny.”
- Bastian, S; Liu, X; Meyerowitz, JT; Snow, CD; Chen, MMY; Arnold, FH. (2011). Engineered ketolacid reductoisomerase and alcoholdehydrogenase enable anaerobic 2-methylpropan-1-ol production a theoretical yield in Escherichia coli. Metabolic Engineering 13: 345 – 352.
- Branon, TC; Bosch, JA; Sanchez, AD; Udeshi, ND; Svinkina, T; Carr, SA; Feldman, JL; Perrimon, N; Ting, AY. (2018). Efficient proximity labeling in living cells and organisms with TurboID. Nature Biotechnology 36: 880 – 887.
- Chen, K; Arnold, FH. (1993). Tuning the activity of an enzyme for unusual environments: sequential random mutagenesis of subtilisin E for catalysis in dimethylformamide. Proceedings of the National Academy of Sciences of the United States of America 90: 5618 – 5622.
- Eigen, M; Gardiner, W. (1984). Evolutionary molecular engineering based on RNA replication. Pure and Applied Chemistry 56: 967 – 978.
- Hult, K; Berglund, P. (2003). Engineered enzymes for improved organic synthesis. Current Opinion in Biotechnology 14: 395 – 400.
- Kłys, A. Słownik łacińsko – polski, polsko – łaciński. Wydanie 1. (2012). Wydawnictwo Level Trading, Czerica.
- Kuchner, O; Arnold, FH. (1997). Directed evolution of enzyme catalysts. Trends in Biotechnology 15: 523 – 530.
- Sandgren, M; Gualfetti, PJ; Shaw, A; Gross, LS; Saldajeno, M; Day, AG; Jones, TA; Mitchinson, C. (2003). Comparison of family 12 glycoside hydrolases and recruited substitutions important for thermal stability. Protein Science 12: 848 – 860.
- Scientifc Background on the Nobel Prize in Chemistry 2018, Stockholm, doi: https://www.nobelprize.org/uploads/2018/10/advanced-chemistryprize-2018.pdf.
- www.atum.bio/company
- www.creative-enzymes.com/
- www.thermofisher.com/pl/en/home/life-science/cloning/gene-synthesis/directed-evolution.html